こんにちは。サーバーエンジニアの佐藤太一(@teach_kaiju)です。
本記事では社内で開発した、数億のデータを処理する仕組みを提供する gem MedPipe を紹介します。
MedPipe とは
「Log のデータを全て取得し、フォーマットして tsv として S3 にアップロードする」という要件があったとします。
この要件を実現するために、例えば以下のような実装を考えることができます。
upload_file_name = "hoge_logs.csv" # 1. S3にアップロードするための file を用意 Tempfile.create do |file| # 2. Log のデータを DB から取得 HogeLog.find_each do |log| # 3. フォーマット処理 formatted_data = format(log) # 4. ファイルに書き込み line = CSV.generate_line(formatted_data, col_sep: "\t") file.puts(line) end # 5. S3にアップロード upload_s3(file, upload_file_name) end def format(log) # 処理 end def upload_s3(file, upload_file_name) # 処理 end
それに対して、MedPipe を使うと以下のように記述できます。
upload_file_name = "hoge_logs.csv" pipeline = MedPipe::Pipeline.new pipeline.apply(PipelineTask::HogeLogReader.new) # 1. Log のデータを DB から取得 .apply(PipelineTask::HogeLogFormatter.new) # 2. フォーマット処理 .apply(MedPipe::PipelineTask::TsvGenerater.new) # 3. ファイルに書き込み .apply(PipelineTask::S3Uploader.new(upload_file_name)) # 4. S3にアップロード pipeline.run
このように、MedPipe を使うことで処理の流れが明確になり、可読性を向上させることができます。
それに加えて以下のような機能を容易に実装することができます。
- 並列処理
- クエリ最適化のための、in_batches を用いない独自データ取得処理
- 件数のカウント
- アップロードするファイルサイズの保存
Ruby エンジニアにとっては Dataflow 等の大規模データ処理ツールと比べて学習コストが低いため、導入を比較的容易に行うことができます。
コンセプト
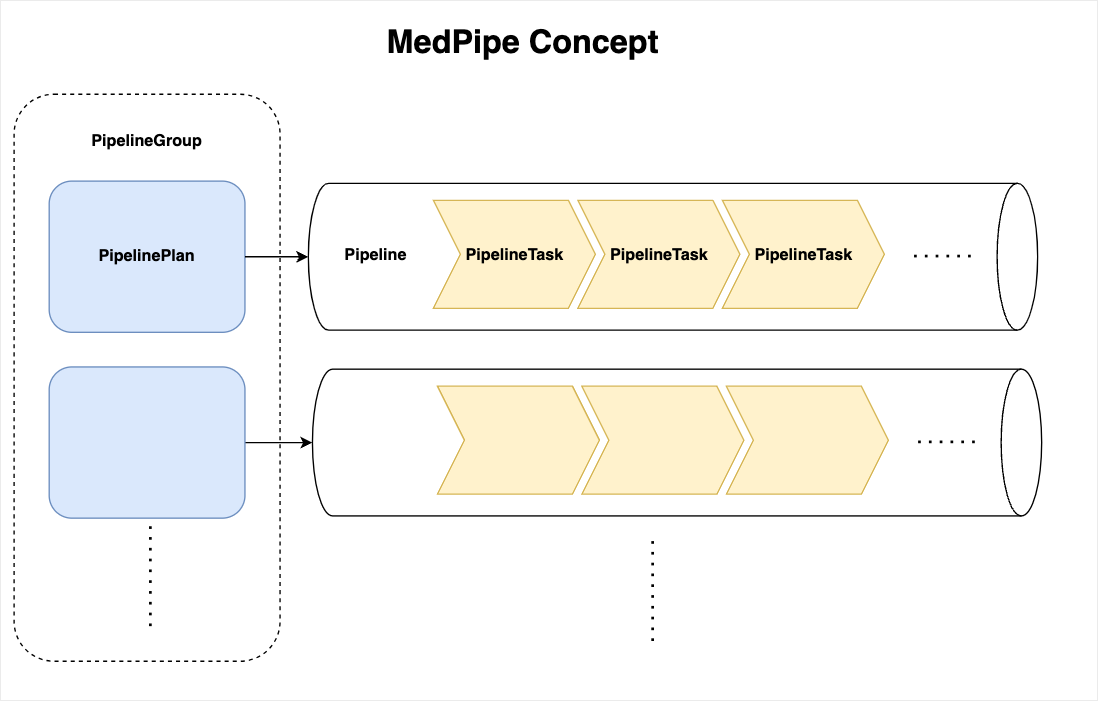
MedPipe では Pipeline に PipelineTask を登録し、それを順番に実行します。
PipelineTask はやりたいことそのものであるため、独自で実装する必要があります。
PipelineTask が実装する必要のあるメソッドは call のみで非常にシンプルです。
def call(context, prev_result) yield "次のTaskの第二引数に渡す値" end
ただし、大量のデータを扱う際には全部のデータをメモリにのせて次の Task に渡すわけにはいきません。
そこで、基本的には Enumerable::Lazy を後続 Task に渡します。
(lazy で Enumerable を Enumerable::Lazy に変換できます)
例
def call(_context, _) yield HogeLog.find_each.lazy end
後続 Task は Enumerable::Lazy を受け取り、map で処理を挟むことで Enumerable::Lazy を維持できます。
def call(_context, records) yield records.map { |record| format_line(record) } end
PipelineTask の他にも PipelineTask を Pipeline に登録する処理など使う準備は必要です。
Usageとサンプルを参考にしてください。
DB からのデータ取得方法
実務で find_each を使う場合には 2 つの問題がありました。
- ActiveRecord のメモリ使用量が多い
- クエリが最適化されない
1 に関しては in_batches + pluck を使うことで解決できますが、2 に関しては解決できません。
参考: Railsでin_batches使うととても遅い
これを解決するために、MedPipe では BatchReader というクラスを開発しました。
使用例:
def call(_context, _) yield MedPipe::BatchReader.new( HogeLog, scope: HogeLog.where(created_at: @target_date.all_day), pluck_columns:, batch_size: BATCH_SIZE ).each.lazy end
これによって find_each のように1件ずつ、pluck_columns で pluck されたデータを後続 Task に渡すことができます。
プロファイリングの仕方
実務では memory_profiler を用いて、以下のようなコードでプロファイリングを行いました。 ※ 執筆にあたり一部修正しています。
module Profiler class << self ... def report(&block) start_time = Time.current result = MemoryProfiler.report(&block) elapsed_time = Time.current - start_time puts "\n\n===== Profiler Report =====" puts "Total allocated: #{bytes_to_mb(result.total_allocated_memsize)} MB (#{result.total_allocated} objects)" puts "Total retained: #{bytes_to_mb(result.total_retained_memsize)} MB (#{result.total_retained} objects)" puts "Elapsed time: #{elapsed_time.round(2)} sec" end ... private ... # bytes to MB 小数点第二位まで def bytes_to_mb(bytes) (bytes / 1024.0 / 1024.0).round(2) end end end
class PipelineTask::Profiler def call(_context, input) Profiler.report do # Lazy の場合、測定するために発火する input.force if input.is_a?(Enumerator::Lazy) yield(input) end end end
pipeline.apply(PipelineTask::Profiler.new)
既存のスクリプトを修正することなく、プロファイリングを行うことができます。
おわりに
本記事では、MedPipe の紹介を行いました。本 gem は弊社初のオープンソースの gem です。
普段様々な OSS のお世話になっているため、提供する側として業界に貢献できることを嬉しく思います。
OSS として世に出すことを許可していただいた会社や一緒に開発した同僚の近藤さん(@tetetratra)に感謝です!
実装が参考になったり、使ってみてよかった場合は、ぜひ MedPipe の GitHub リポジトリにスターをいただけると励みになります。
是非読者になってください!
メドピアでは一緒に働く仲間を募集しています。 ご応募をお待ちしております!
■募集ポジションはこちら medpeer.co.jp
■エンジニア紹介ページはこちら engineer.medpeer.co.jp
■メドピア公式YouTube www.youtube.com
■メドピア公式note
style.medpeer.co.jp